



Artículos originales
Determinação de amido e carboidrato em folhas de mangueira com o uso espectroscopia Vis-NIR.
Determinación de almidón y carbohidrato en hojas de mango mediante espectroscopía Vis-NI
Determination of starch and carbohydrate in mango leaves using Vis-NIR spectroscopy
Temas Agrarios
Universidad de Córdoba, Colombia
ISSN: 0122-7610
ISSN-e: 2389-9182
Periodicidade: Semestral
vol. 27, núm. 2, 2022
Recepção: 14 Dezembro 2022
Aprovação: 20 Dezembro 2022

Resumo: A produção de manga apresenta desafios, como a maturação dos ramos da mangueira, que aliada a boa nutrição e bioquímicos envolvidos nesse processo como o carboidrato e o amido favorece o desenvolvimento do vegetal. A utilização de técnicas não destrutivas e rápidas para determinar os teores desses componentes na planta, como a espectroscopia, pode otimizar a realização das análises desses componentes. Diante disso, este trabalho teve como objetivo desenvolver modelos preditivos para determinação de teores de amido e carboidratos em folhas de mangueira “Palmer” com o uso da espectroscopia Vis-NIR submetidas a diferentes fontes de potássio. O trabalho foi desenvolvido na região do Vale do São Francisco, seguindo as seguintes etapas: (1) a amostragem das folhas; (2) análise espectrais; (3) determinação em laboratório dos teores de carboidratos e amido; e (4) desenvolvimento dos modelos preditivos de regressão e classificação. Os modelos preditivos de regressão utilizados foram a Regressão por Componentes Principais (PCR) e a Regressão por Quadrados Mínimos Parciais (PLSR). Também foram desenvolvidos modelos discriminantes supervisionado para classificar as folhas da mangueira de acordo com as diferentes fontes potássicas utilizada, utilizando a análise discriminante linear (LDA). A espectroscopia Vis-NIR apresentou valores baixos para a avaliação não destrutivas de folhas de mangueira “Palmer” utilizando PCR e PLSR para predição de carboidrato e amido com R2 de 0,58 menor que os modelos considerados excelentes (R2 >0,90); O desenvolvimento de modelos de classificação não possibilitou a discriminação das diferentes fontes de potássio em folhas de mangueira “`Palmer” com precisão de 64,2%.
Palavras-chave: Análise bioquímicas, Adubos potássicos, Mangífera indica L, Métodos não destrutivos, Quimiometria.
Resumen: La producción de mango presenta desafíos, como la maduración de las ramas del mango, que, combinado con una buena nutrición y bioquímicos involucrados en ese proceso, como los carbohidratos y el almidón, favorecen el desarrollo de la planta. La utilización de técnicas no destructivas y rápidas para determinar los niveles de estos componentes en la planta, como la espectroscopia, puede optimizar el análisis de esos componentes. Por lo tanto, este trabajo tuvo como objetivo desarrollar modelos predictivos para la determinación de contenido de almidón y carbohidratos en hojas de mango “Palmer” mediante espectroscopía Vis-NIR sometidas a diferentes fuentes de potasio. El trabajo se llevó a cabo en la región del Valle de São Francisco, siguiendo las siguientes etapas: (1) muestreo de hojas; (2) análisis espectral; (3) determinación de laboratorio de contenido de carbohidratos y almidón, y (4) desarrollo de los modelos predictivos de regresión y clasificación. Los modelos de regresión predictiva utilizados fueron Regresión de Componentes Principales (PCR) y Regresión de Mínimos Cuadrados Parciales (PLSR). También se desarrollaron modelos discriminantes supervisados para clasificar las hojas de mango según las diferentes fuentes de potasio utilizadas, utilizando análisis discriminante lineal (LDA). La espectroscopía Vis-NIR mostró valores bajos para la evaluación no destructiva de hojas de mango “Palmer” usando PCR y PLSR para predicción de carbohidratos y almidón con R2 de 0,58 inferior a los modelos considerados excelentes (R2 >0.90); El desarrollo de modelos de clasificación no permitió las diferentes fuentes de potasio en hojas de mango “`Palmer” con una precisión de 64,2%.
Palabras clave: Análisis bioquímicos, Abonos potásicos, Mangifera indica, Métodos no destructivos, Quimiometría.
Abstract: Mango production presents challenges, such as the maturation of the mango branches, which, combined with good nutrition and biochemicals involved in this process, such as carbohydrate and starch favor the development of the plant. Therefore, the use of non-destructive, fast techniques to determine the levels of these components in the plant, such as spectroscopy, can optimize the analysis of these components. Therefore, this work aimed to develop predictive models for determination of starch and carbohydrate contents in “Palmer” mango leaves using vis-nir spectroscopy subjected to different potassium sources. The work was carried out in the region of San Francisco Valley, using the following steps: (1) leaf sampling; (2) spectral analysis; (3) lab determination of carbohydrate and starch contents; and (4) development of predictive regression and classification models. The predictive regression models used were Principal Components Regression (PCR) and Partial Least Squares Regression (PLSR). Supervised discriminant models were also developed to classify mango leaves according to different potassium sources used, using linear discriminant analysis (LDA). Vis-NIR spectroscopy showed low values for the non-destructive evaluation of “Palmer” mango leaves using PCR and PLSR for carbohydrate and starch prediction with R2 of 0.58 lower than the models considered excellent (R2 >0.90); The development of classification models did not allow the discrimination of different sources of potassium in “Palmer” mango leaves with an accuracy of 64.2%.
Keywords: Biochemical analysis, Chemometrics, Mangífera indica L, Non-destructive methods, Potassium fertilizers.
INTRODUÇÃO
Pertencente à família Anacardiaceae, a mangueira (Mangífera índica L.) é uma frutífera de grande importância para diversos países, como o Brasil, em que a maior parte da produção de manga está localizada na região do Vale do São Francisco que se concentra no Nordeste brasileiro, onde são produzidas principalmente mangas das cultivars ‘TommyAtkins’ e ‘Palmer’ (Souza e Lima, 2023).
Mas assim como para qualquer outra cultura, existes empecilhos que precisam ser superados ou controlado como influência de fatores ambientais, desuniformidade no florescimento e maturação dos ramos (Oliveira et al., 2019). Entre esses fatores, a maturação dos ramos da mangueira é importância para a cultura da mangueira, principalmente no período vegetativo, que é a etapa que antecede a floração, pois nessa fase que será definida a arquitetura da planta, sendo responsável pelo bom florescimento e consequentemente produção de frutos com qualidade e produtividade (Cavalcante et al., 2018).
Os níveis de substâncias de reserva presentes no ramo da mangueira, como carboidratos e amido, são vitais para um bom amadurecimento dos ramos. Teores adequados desses componentes na folha da mangueira estão envolvidos diretamente na maturação dos ramos e posteriormente na intensidade e uniformidade do florescimento (Oliveira et al., 2018). Mas as análises para determinação de carboidratos e amido são onerosas, destrutivas e demanda muito tempo para processa-la, como por exemplo o método de Dubois et al. (1956), na qual exigem o uso de reagentes caros e, além disso, alguns apresentam seu uso restrito, como o ácido sulfúrico.
Uma tecnologia que veem ganhando notória divulgação é o uso de Espectroscopia Vis-NIR, uma técnica não destrutiva que pode ser utilizada para descrever inúmeros atributos de uma planta como por exemplo: Determinação de carboidrato solúvel, amido e sacarose (Souza, 2022; Frey et al., 2020; Nkajima et al., 2019; Das et al., 2018; De Bei et al., 2017), pigmentos fotossintéticos e teor de clorofila (Oliveira et al., 2021), teor de matéria orgânica no solo (González-Aguiar et al., 2020) e também analise qualidade de diversos produtos, como por exemplo, analise de vinho (Barreto, 2020; Genisheva et al., 2018) maturação e qualidade de frutos (Souza et al., 2022).
Diante do exposto, o objetivo desse trabalho foi determinar teores de amido e carboidrato com o uso da espectroscopia Vis-NIR em folha de mangueira “Palmer” submetidas às fontes de potássio no período vegetativo.
MATERIAL E MÉTODOS
Para uma melhor organização do experimento, este foi dividido em quatro etapas principais, que consistiram em: (1) amostragem foliares; (2) análises espectrais; (3) determinação em laboratório de carboidrato e amido; (4) quimiometria - modelos de regressão e classificação.
Seguindo a sequência, após a etapa 1, amostragem, as folhas da mangueira foram submetidas às análises espectrais para aquisição dos espectros de absorbância, e logo em seguida, às análises de referência, que são as análises convencionais de determinação dos compostos bioquímicos de carboidrato e amido foliar; os dados obtidos através de procedimento espectrais passaram por uma série de pré-tratamentos, cujo objetivo foi a remoção de informações irrelevantes, que não contribuem ou diminuem o desempenho dos modelos e após o pré-tratamento dos dados, métodos de regressão foram aplicados, para o desenvolvimento dos modelos de predição.
(1) Amostragem das folhas
O trabalho foi densenvolvido na fazenda experimental da Universidade do Vale do São Francisco – UNIVASF, que se localiza no Vale do São Francisco, especificamente no Submédio, na cidade de Petrolina-PE. Região que apresenta característica climática semiárida quente com baixa ocorrência de precipitações (Souza et al., 2020).
Mangueiras cv. ‘Palmer’ no estágio de desenvolvimento vegetativo foram utilizadas para a coletada de suas folhas na altura mediana da copa da planta, na região do último fluxo vegetativo maduro. As coletas foram realizadas após a poda de formação, sendo realizadas 4 podas de formação, o período entre uma poda e outra durou cerca de 3 a 5 meses. A cada coleta obteve-se um total de 28 amostras, que foram retiradas de plantas previamente tratadas com diferentes fontes de potássio (K): 7 plantas pulverizada com (cloreto de potássio (KCl); 7 plantas com sulfato de potássio (K SO ) amostras foram descongeladas até atingirem a temberatura ambiente.
Cada amostra correspondeu a um conjunto total de 7 folhas com parâmetros iguais (tamanho, forma e cor), o qual foi feita a leitura espectral e feita uma média, determinando assim o valor de uma amostra.
(2) Análises espectrais
O equipamento utilizado foi o sistema de espector de refletância composto pelos seguintes compontentes: espectrômetro FieldSpec3 (Analytical Spectral Devices, Boulder, Colorado, USA) que opera na faixa de comprimento de onda de 350 a 2500 nm, com resolução de 3 a 10 nm e tempo por varredura de 100 ms, equipado com probe plant; e computador com o software RS3 (Analytical Spectral Devices, Boulder, Colorado, USA). A leitura espectraias ocorreu no limpo foliar, sendo o sistema fígado para aquecimento 30 minutos antes das aquisições dos espectros.
Cada um dos espectos é composto por cerca 30 varreduras que são executadas pelo equipamento.A probe plant possui uma referência branca, com a qual as operações de calibração são implementadas antes da aquisição dos dados. Os valores de refletância foram transformados em medições de absorbância (log (1/R)), onde R significa refletância, utilizando o software ViewSpec Pro (Analytical Spectral Devices, Boulder, Colorado, USA).
(3) Determinação em laboratório de carboidrato e amido
Feitas as medições de refletância, foi determinado as análises bioquímicas de referência para determinação dos teores de carboidratos solúveis totais (mmol g-1 de MF) e amido (μg g-1 de MF) que foram extraídas das amostras foliares.
Para determinação do teor de carboidrato solúveis totais foi utilizado a metodologis de Dubois et al. (1956). Consistiu na extração de 1 g de material foliar fresco, homogeneizado em 4 ml de solução tampão fosfato de potássio monobásico, 0,1 M e pH 7,0. Após centrifugação e banho-maria, foram colocados em tubos de ensaio 10 μl do sobrenadante (após teste de diluição),490 μl do tampão fosfato monobásico, 500 μl de fenol e 2,5 ml de ácido sulfúrico, seguido de repouso e posteriormente agitação, e depois novamente deixados em uma bandeja contendo água à temperatura ambiente (25ºC) por 10 minutos. Sendo por fim, realizadas as leituras em um espectrofotômetro a 490 nm.
O teor de amido foi determinado segundo Neves e Morais (2005). Para a extração, utilizou-se 0,05 g de material foliar fresco, homogeneizado em 4 ml de H2SO4 (ácido sulfúrico). Foram tomadas alíquotas de 0,5 ml de extrato: a cada alíquota foi adicionado 0,95 ml de água e 3 ml de antrona. Após o preparo das amostras, foram realizadas as leituras em um espectrofotômetro a 620 nm.
(4) Quimiometria - modelos de regressão e classificação
Após os dados espectrais serem gerados, estes foram submetidos ao pré-processamento filtro de média móvel (Moving Average, MA) e correção de sinal ortogonal (Orthogonal Signal Correction, OSC).
A partir das matrizes pré-tratadas foram desenvolvidos os modelos de Regressão pelo método das componentes principais (PCR) e regressão pelo método dos quadrados mínimos parciais (PLSR). Para isso, os dados brutos ou pré-tratados foram utilizados como variáveis de entrada independentes (X) e os atributos de interesse (amido, carboidrato) como variáveis de entrada dependentes (Y). Foi aplicada a v alidação cruzada em todas as amostras dos conjuntos de dados e o número ótimo de fatores foi definido através do menor valor obtido do erro quadrático médio da raiz da validação cruzada (RMSECV). O algoritmo utilizado para a construção dos modelos foi NIPALS (Geladi e Kowalski, 1986), com 100 interações.
Após processamento, foram selecionados os melhores modelos para cada atributo bioquímico foliar, em suas respectivas faixas espectrais. Posteriormente, foi feita a seleção de variáveis através de uma PCA e exclusão das variáveis com menor influência e não significativas (p-valor > 0,05) para os modelos através do método Jack-Knife (Martens, 2000). Foram selecionados os comprimentos de ondas associados aos picos de absorbâncias em todas as componentes principais selecionadas, seguido da remoção das variáveis com menores coeficientes de regressão. O modelo foi recalculado a cada variável removida, de modo que, o objetivo foi encontrar modelos com os melhores parâmetros de desempenho e menor número de variáveis e que todas sejam significativas para a construção dos modelos. Os comprimentos de onda selecionados a partir dos picos de absorbâncias e aquelas submetidos à ANOVA e teste de significância a 5%.
O desempenho dos modelos foi avaliado a partir da comparação dos parâmetros estatísticos, a saber: coeficiente de determinação (R2); erro quadrático médio de calibração (RMSEC); erro padrão de calibração (SEC); erro quadrático médio de validação cruzada (RMSECV); erro padrão de validação cruzada (SECV). Estes parâmetros são definidos pelas equações de (1) a (5):
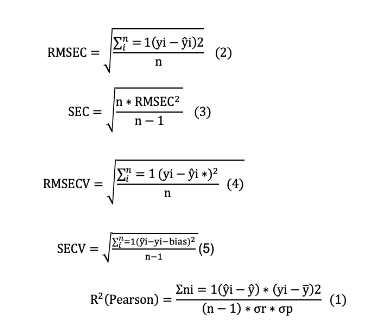
Onde ŷi* é o valor previsto pelo modelo de calibração; ŷi* é o valor previsto pelo modelo na etapa de validação cruzada; yi é o valor de referência; ŷ é a média dos valores de predição; n é o número de amostras na calibração ou validação; m é o número de amostras previstas; σr é o desvio-padrão dos valores de referência; σp é o desvio-padrão dos valores de predição.
Os melhores modelos são aqueles que apresentam os maiores valores de coeficiente de determinação da validação cruzada (R²cv), seguido dos menores valores de erro quadrático médio de validação cruzada (RMSECV).
Logo em seguida foi utilizado a análise de componentes principais (PCA) para explorar o possível agrupamento de amostras e fornecer informações sobre estrutura latente da matriz espectral. A matriz de dados foi composta das folhas da mangueira “Palmer” nas diferentes datas de coletas levando em consideração os diferentes fertilizantes potássicos utilizados no experimento.
Identificados os agrupamentos entre as diferentes coletas foliares utilizando o PCA, modelo discriminante supervisionado foi utilizado para classificar as folhas da mangueira de acordo com os diferentes fertilizantes aplicados, utilizando a análise discriminante linear (LDA).
Um conjunto de treinamento e teste de amostras compostas por espectros de folhas aonde foram aplicadas previamente três fertilizantes potássico (cloreto de potássico, sulfato de potássio e Speed folll) mais uma testemunha, classificadas segundo Coombe (1995), foram utilizados para a confecção dos modelos matemáticos. A classificação foi realizada para cada folha amostra. A matriz de dados foi constituída por valores de absorbância, sendo esta variável independente, e por classe definida, como variável dependente.
O método LDA foi utilizado com diferentes técnicas para o cálculo das distâncias lineares entre amostras, e suas consequentes similaridades. Sendo utilizado o espectro adquirido a (350 a 2500 nm).
O desempenho do modelo de classificação supervisionados usando matriz de confusão. Os valores de precisão (Prec), sensibilidade (Sen), seletividade (Sel) e taxa de falsos positivos (TFP) foram obtidos a partir das matrizes de confusão de cada modelo, conforme equação de 6 a 9:

Em que VP, VN, FP e FN são os valores positivos, verdadeiro negativo, falso positivo e falso negativo, respectivamente. As Em que VP, VN, FP e FN são os valores positivos, verdadeiro negativo, falso positivo e falso negativo, respectivamente. As medidas gerais de desempenho, ou seja, precisão, sensibilidade, seletividade e taxa de falso positivo, foram calculadas com base nas medidas de desempenho.
RESULTADOS E DISCUSSÃO
Espectro: análise exploratória
Na figura 1, observase o espectro médio bruto de absorbância das folhas da mangueira “Palmer” para os diferentes fertilizantes potássicos aplicados. Esses espectros apresentaram quatro principais pontos de absorção nas regiões, sendo eles uma faixa estável de absorção em torno de 460 nm, além dos picos próximos dos comprimentos de 670, 1450 e 1930 nm, como mostram os espectros médios dos dados. Verificase que ambos os fertilizantes potássicos obtiveram o mesmo comportamento com pontos de absorção muito próximos.
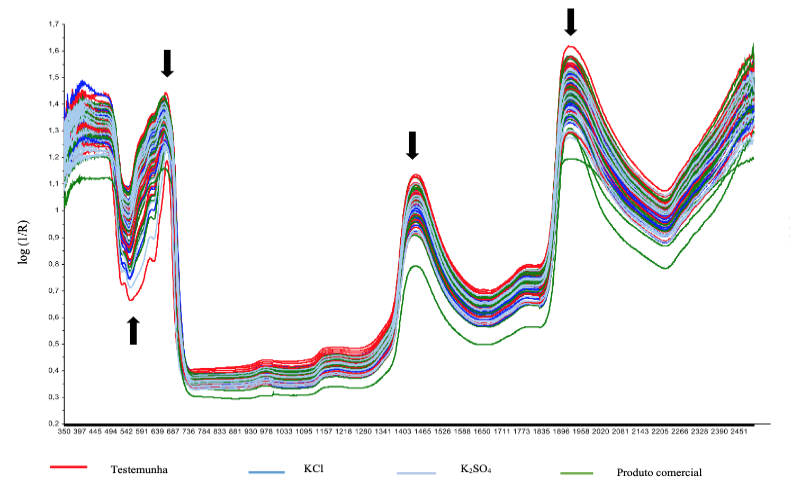
Como observado na figura 1, os picos iniciais se encontram em torno de 460 a 670 nm, e isso pode estar associa a presença de clorofila nas folhas. Sendo que, a clorofila “a” apresenta absorção entre 450 e 680 nm, a clorofila “b” em 475 e 675 nm (Gould et al., 2018). Um exemplo foi o trabalho proposto por Novo, 2010, na qual foi observado que em uma folha verde sadia, há uma alta absorbância de luz no comprimento de 460 e 670 nm na região do visível devido às clorofilas. E os comprimentos de onda de 1450 e 1930 nm é devido o conteúdo de água na folha. Sendo que, para De Bei et al. (2017) esses comprimentos de onda podem estar relacionados aos teores de açúcares e amido na folha.
A estrutura interna, composição e morfologia de uma folha influencia no comportamento espectral e apresenta diferença se forem de grupos geneticamente diferente. E esse comportamento é semelhante ou parecido entre todas as folhas de plantas no espectro Vis-NIR (350-2500 nm) e exerce influência principal da incidência da luz com a clorofila e ligações moleculares de N-H, O-H, C-H (Curran, 1989).
Modelos de regressão PCR e PLSR
Foram desenvolvidos modelos PCR e PLSR para o conjunto de dados com folhas de mangueira para os atributos carboidrato e amido. A tabela 1 apresenta os melhores resultados para cada modelo analisado nas diferentes datas de coleta. Para a variável amido, tanto para o PCR quanto para o PLSR, o melhor modelo de predição foi da coleta 1, que apresentaram R2cv de 0,58 e RMSECV de 0,03 iguais.
| Modelo | Coleta | Fatores | R2 cal | RMSEC | SEC | R2 cv | RMSECV | SECV |
| amido | ||||||||
| PCR | 1 | 1 | 0,59 | 0,03 | 0,03 | 0,58 | 0,03 | 0,03 |
| PCR | 2 | 1 | 0,15 | 0,07 | 0,07 | 0,10 | 0,07 | 0,07 |
| PCR | 3 | 2 | 0,39 | 0,03 | 0,03 | 0,34 | 0,04 | 0,04 |
| PCR | 4 | 3 | 0,33 | 0,04 | 0,04 | 0,18 | 0,05 | 0,05 |
| PLSR | 1 | 1 | 0,59 | 0,03 | 0,03 | 0,58 | 0,03 | 0,03 |
| PLSR | 2 | 1 | 0,15 | 0,07 | 0,07 | 0,10 | 0,07 | 0,07 |
| PLSR | 3 | 3 | 0,81 | 0,02 | 0,02 | 0,38 | 0,03 | 0,04 |
| PLSR | 4 | 1 | 0,15 | 0,05 | 0,05 | 0,07 | 0,05 | 0,05 |
| carboidrato | ||||||||
| PCR | 1 | 3 | 0,42 | 15,14 | 15,42 | 0,32 | 16,92 | 17,23 |
| PCR | 2 | 2 | 0,30 | 12,06 | 12,28 | 0,21 | 13,25 | 13,49 |
| PCR | 3 | 1 | 0,13 | 31,03 | 31,60 | 0,01 | 34,31 | 3493 |
| PCR | 4 | 0 | -- | -- | -- | -- | -- | -- |
| PLSR | 1 | 2 | 0,56 | 13,15 | 13,39 | 0,28 | 17,48 | 17,80 |
| PLSR | 2 | 1 | 0,23 | 12,66 | 12,90 | 0,18 | 13,52 | 13,77 |
| PLSR | 3 | 1 | 0,13 | 30,96 | 31,53 | 0,02 | 34,19 | 34,81 |
| PLSR | 4 | 0 | -- | -- | -- | -- | -- | -- |
Em relação a variável carboidrato, o melhor modelo PCR foi da coleta1, mas apresentando R2cv de 0,32 muito abaixo do amido e RMSECV de 16,9 bastante elevada. E para o modelo PLSR apresentou R2cv de 0,28 e RMSECV de 17,48 para a coleta1, onde foi possível observar que as diferentes condições de coletas das variáveis amido e carboidrato, exerce influência na confecção dos modelos dos conjuntos de dados (Tabela 1).
O que determina os melhores modelos são aqueles que apresentam os maiores valores de coeficiente de determinação de validação cruzada (R²cv) seguido de valores baixos de erro quadrático médio de validação cruzada (RMSECV). E no presente estudo, em relação ao amido e carboidrato, o desempenho do modelo desenvolvido foi baixo, semelhante ao trabalho de Souza, (2022), que para a variável carboidrato solúvel obteve R2CV de 0,39, sendo encontrado no presente experimento R2cv de 0,32. Diferentes de outros estudos, como de De Bei et al. (2017) e de Lohr et al. (2017), que obtiveram respectivamente R2cv de 0,86 e 0,83 valores bem melhores. Coeficiente de determinação superior a 0,9 são considerados excelentes, pois explica melhor que a variabilidade dos dados de saída ser explica pela variabilidade das variáveis de entrada.
Entretanto, apesar dos autores já citados apresentarem melhores desempenhos em razão de obterem melhores modelos com R2cv elevado, os mesmos utilizaram pré-tratamento utilizando SNV (Standar Normal Variate Transformation) (Barnes; Dhanoa; Lister, 1989) que proporciona modelos inferiores aos utilizado neste experimento, que foram pré-tratados com OSC. Além disso, foi utilizado um maior número de amostras (578 amostras) que no presente estudo (112 amostras), sendo que, o mesmo ocorreu como o trabalho de Souza, (2022) que obteve resultado parecido com o estudo em questão.
Na tabela 2, observa-se o desempenho dos modelos PCR e PLSR com todas as coletas juntas com espectro completo (350-2500 nm) e com seleção de comprimento de onda, após a aplicações de técnicas de pré-tratamento.
| Modelo | Coleta | Fatores | R2cal | RMSEC | SEC | R2cv | RMSECV | SECV |
| amido | ||||||||
| PCR | completo | 2 | 0,35 | 0,05 | 0,05 | 0,33 | 0,05 | 0,05 |
| PLSR | completo | 15 | 0,96 | 0,01 | 0,01 | 0,47 | 0,04 | 0,04 |
| PCR | seleção | 1 | 0,32 | 0,05 | 0,05 | 0,31 | 0,05 | 0,05 |
| PLSR | seleção | 6 | 0,68 | 0,03 | 0,03 | 0,49 | 0,04 | 0,04 |
| carboidrato | ||||||||
| PCR | completo | 1 | 0,56 | 26,49 | 26,61 | 0,56 | 26,95 | 27,07 |
| PLSR | completo | 2 | 0,72 | 21,23 | 21,33 | 0,58 | 26,19 | 26,31 |
| PCR | seleção | 1 | 0,56 | 26,49 | 26,61 | 0,56 | 26,95 | 27,07 |
| PLSR | seleção | 3 | 0,71 | 21,36 | 21,46 | 0,67 | 23,20 | 23,30 |
Para o conjunto de dados da variável “amido” o melhor modelo encontrado foi o PLSR-seleção, com R2cv de 0,49 e RMSECV de 0,04. Já para a variável “carboidrato”, houve um comportamento melhor, com R2cv de 0,67 e RMSECV de 23,20 também para o modelo PLSR de seleção.
Nota-se que os melhores modelos reconstruídos com seus respetivos pré-tratamentos matemáticos foram aqueles obtidos com espectro de seleção de variável (Tabela 2). Diferente do encontrado por Frey et al. (2020), que obteve R2cv de 0,56 em modelos PLSR com todos os comprimentos de onda, ou seja, modelos completos, e R2cv de 0,18 com seleção de variáveis. Segundo Wu et al., 2010 determinados comprimentos de ondas têm pouca ou nenhuma contribuição sobre os modelos desenvolvidos. Entretanto, observase que no presente experimento, a seleção de determinado comprimento de onda contribuiu para aquisição de melhores modelos matemáticos. Sendo que, os modelos de variáveis selecionadas, em comparação com os modelos com variável completo, apresentam entradas reduzidas, o que minimiza o tempo de calibração dos modelos (Cao et al., 2010).
Ademais a seleção de variáveis é uma excelente ferramenta para melhorar o desempenho do modelo, aumentar a precisão das variáveis selecionadas e aumentar a compreensão e interpretação dos modelos obtidos (Yun et al., 2019).
Pode-se notar também a superioridade do modelo PLSR em relação ao PCR, sendo que, o PLSR é o método recomendado para medições de amostras multivariadas, devido a sua capacidade de trabalhar com um grande número de amostras em problema de colinearidade (Genisheva et al., 2018;Paz-Kagan et al., 2020).
Em seguida, segue abaixo na figura 2, os gráficos de dispersão e validação cruzada dos modelos PLSR que foram confeccionados com as assinaturas espectrais.
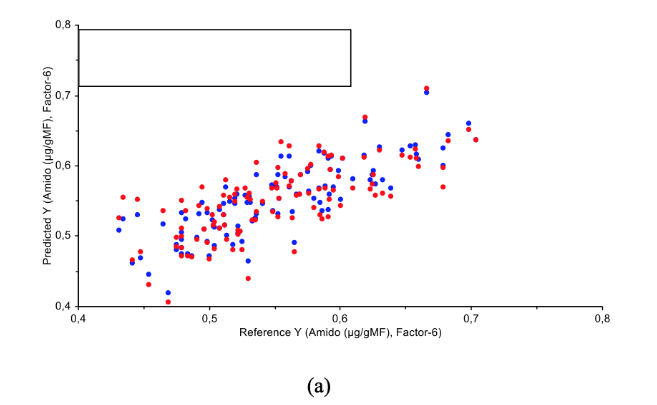
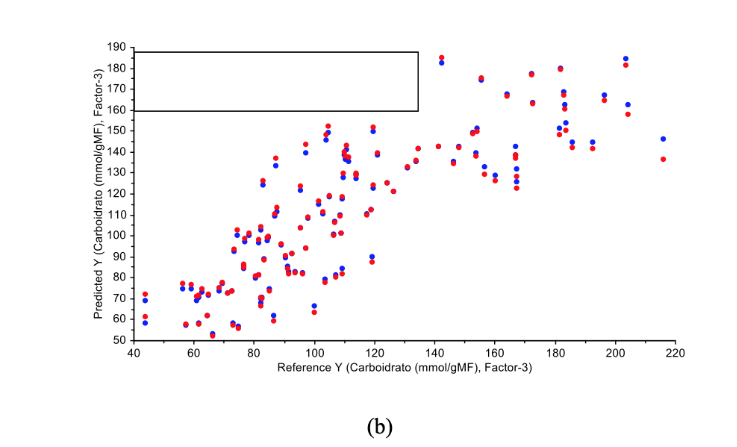
Pode-se notas que os modelos PLSR construídos com as assinaturas espectrais apresentaram baixa correlação tanto para a variável amido (R2=0,68), quanto para o carboidrato (R2=0,71). Contudo essa inferioridade não compromete a atividade de previsão dos atributos de qualidade (Costa et al., 2019).
Os melhores modelos preditivos são obtidos a partir das assinaturas espectrais das bandas espectrais analisadas, com isso é possível determinar os teores de carboidrato e amidos sem que aja novas análises em laboratório considerando a precisão de cada modelo.
Método supervisionado de classificação (LDA)
Para poder discriminar as diferentes fontes de potássio utilizado no experimento com o uso de espectroscopia VIS-NIR para as diferentes coletas, foi utilizado o método de classificação supervisionado como mostrada na tabela 3.
| Coleta | Exatidão | Sensibilidade | Seletividade | TFP |
| 1 | 64,28% | 64,28% | 83,19% | 16,80% |
| 2 | 50,00% | 50,00% | 76,81% | 23,18% |
| 3 | 60,71% | 60,71% | 73,47% | 26,52% |
| 4 | 60,71% | 60,71% | 77,76% | 22,23% |
Nota-se que, o modelo classificatório utilizado não foi capaz de prever com uma exatidão elevada as diferentes fontes de potássio nas diferentes datas de coleta, sendo que, para a coleta 1 o LDA com distância calculada pelo método linear apresentado maiores taxas de acerto de que as demais coletas, com exatidão de 64,284% (Tabela 3).
CONCLUSÕES
A espectroscopia Vis-NIR apresentou valores baixos para a avaliação não destrutivas de folhas de mangueira “Palmer” utilizando PCR e PLSR para predição de carboidrato e amido com R2 de 0,58 menor que os modelos considerados excelentes (R2>0,90).
O desenvolvimento de modelos de classificação não possibilitou a discriminação das diferentes fontes de potássio em folhas de mangueira “Palmer” com precisão de 64,2%.
REFERENCIAS
Barnes, R. J., dhanoa, M. S., Lister, S. J. 1989. Standard Normal Variate Transformation and De Trending of Near-Infrared Diffuse Reflectance Spectra. Appl Spectrosc 43(5): 772–777. http://doi/10.1366/0003702894202201
Barreto, N. S. 2020. Aplicação da técnica de espectroscopia do visível e infravermelho próximo (Vis/NIR) no controle físico-quí- mico da qualidade de vinhos produzidos no Submédio do Vale do São Francisco. Dissertação Mestrado em Ciência e Tecnologia de Alimentos, Universidade Federal de Sergipe- UFSE, São Cristovão.
Cao, F., Wu, D., He,Y. 2010. Soluble solids contente and Ph prediction and varieties discrimination of grapes based on visible-near infrared spectroscopy. Comput. Electron. Agric, 71(6): 15-18. http://dx.doi.org/10.1016/j.compag.2009.05.011
Cavalcante, Í. H. L., Santos, G. N. F., Dos.,Silva, M. A. Da., Martins, R. S., Lima, A. M. N., Modesto, P. I. R., Alcobia, A.M., SILVA, T. R. S., Araujo, E. A. R. A., Beckmann-Cavalcante, M. A. 2018. New approach to induce mango shoot maturation in Brazilian semi-arid environment. Journal of Applied Botany and Food Quality, 91: 281-286. https://doi. org/10.5073/JABFQ.2018.091.036
Coombe, B. G. 1995. Adoption of a system for identifying grapevine growth stages. Australian Journal of Grape and Wine Research, 1(2): 100-110. https://doi/10.1111/j.1755-0238.1995.tb00086.x
Costa, D. S., Mesa, N. F. O., Freire, M. S., Ramos, R. P., Medeiros, B. J. T. 2019. Development of predictive models for quality and maturation stage atributes of wine grapes usin vis-nir reflectance spectroscopy. Posthavest Biology and Technology, 150: 166-178. https://doi.org/10.1016/j.postharvbio.2018.12.010
Curran, P. J. 1989. Remote sensing of foliar chemistry. Remote Sensing of Environment, 30(3): 271–278. https://doi. org/10.1016/0034-4257(89)90069-2
Das, B., Sahoo R. N., Pargal S., Krishna G., Verma R., Chinnusamy V., Sehgal V. K., Gupta V. K., Traço S. K., Swain P. 2018. Quantitative monitoring of sucrose, reducing sugar and total sugar dynamics for phenotyping of water-deficit stress tolerance in rice through spectroscopy and chemometrics. Spectrochimica Acta Part A: Molecular and Biomolecular Spectroscopy, 192(5): 41–51. https://doi.org/10.1016/j. saa.2017.10.076
De Bei, R., Fuentes, S., Sullivan, W., Edwards, E. J., Cozzolino, D. 2017. Rapid measurement of total non-structural carbohydrate concentration in grapevine trunk and leaf tissues using near infrared spectroscopy. Computers and Electronics in Agriculture, 136(15):176–183. https:// doi.org/10.1016/j.compag.2017.03.007
Dubois, M., Gilles, K. A., Hamilton, J. K., Rebers, P. A., Smith, F. 1956. Colorimetric Method for Determination of Sugars and Related Substances. Anaytical Chemistry, 28(3): 350-356. https://pubs.acs.org/doi/10.1021/ac60111a017
Frey, L. A., Baumann, P., Aasen, H., Studer, B., Kolliker, R. 2020. A Non-destructive Method to Quantify Leaf Starch Content in Red Clover. Frontiers in Plant Science, 11. https://doi.org/10.3389/fpls.2020.56994
Geladi, P., Kowalski, B. R. 1986. Partial least-squares regression: a tutorial. Anal. Chimica Acta, 185:1-17, 1986.
Genisheva, Z., Quintelas, C., Mesquita, D. P., Ferreira, E. C., Oliveira, J. M., Amaral, A. L. 2018. New PLS analysis approach to wine volatile compounds characterization by near infrared spectroscopy (NIR). Food Chemistry, 246: 172–178. https://doi.org/10.1016/j.foodchem.2017.11.015
Gonzáles-Aguiar, D., Colás-Sánchez, A., Rodriguez-López, O., Álvarez-Vázquez, D. L., Gattorno-Muñoz, S., Chacón-Iznaga, A. 2020. Estimación De La Materia Orgánica En Suelo Pardo Mullido Medianamente Lavado Mediante Espectroscopia Vis-NIR. Centro Agrícola, 23-32. http://scielo.sld. cu/scielo.php?script=sci_art text&pi- d=S0253-57852020000300023&ln-g=es&nrm=iso
Gould, K., Jay-Allemand, C., Logan, B. A., Baissac, Y., Bidel, L. P. R. 2018. When are foliar anthocyanins useful to plants? Re-evaluation of the photoprotection hypothesis using Arabidopsis thaliana mutants that differ in anthocyanin accumulation. Environmental and Experi- mental Botany, 154(1): 11-22. https://doi. org/10.1016/j.envexpbot.2018.02.006
Lohr, D., Tillmann, P., Druege, U., Zerche, S., Rath, T., Meinken, E. 2017. Non-destructive determination of carbohydrate reserves in leaves of ornamental cuttings by near-infrared spectroscopy (NIRS) as a key indicator for quality assessments. Biosys- tems Engineering, 158:51–63. https://doi.org/10.1016/j.biosystemseng.2017.03.005
Martens, H., Martens, M. 2000. Modified Jack-knife estimation of parameter uncertainty in bilinear modelling by partial least squares regression (PLSR). Food Quality and Preference, 11(1): 5–16. https://doi.org/10.1016/S0950-3293(99)00039-7
Nakajima, S., Shiraga, K., Suzuki, T., Kondo, N., Ogawa, Y. 2019. Quantification of starch content in germinating mung bean seedlings by terahertz spectroscopy. Food Chemistry, 294(1): 203–208. https://doi.or- g/10.1016/j.foodchem.2019.05.065
Neves, L., Moraes, D. M. 2005. Vigour And A-Amylase Analisis In Seeds Of Rice Cultivars Submitted To Several Treatments With Acetic Acid. Revista de Ciências Agroveterinárias, 4(1): 35–43.
Novo, E. M. L. M. 2010. Sensoriamento remoto: princípios e aplicações. 3a edição ed. São Paulo: Blucher.
Oliveira, G. P., Siqueira, S. L., Cecon, P. R., Salomão, L. C. C. 2018. Teores de carboidratos em mangueira “Ubá” submetida a diferentes doses de paclobutrazol. Revista de Ciências Agrárias, 41(3): 749–756. https://doi.org/10.19084/RCA18016
Oliveira, M. B., Figueiredo, M. G. F., Pereira, M. C. T., Mouco, M. A. Do C., Ribeiro, l. M., Simões, M. O. M. 2019. Structural and cytological aspects of mango floral induction using paclobutrazol. Scientia Horticulturae, 262(27): 109057. https://doi.org/10.1016/j.scienta.2019.109057
Oliveira, N., Tinini, R., Costa, D. S., Ramos, R., Wetterich, C., Teruel, B. 2021. Predictive models of chlorophyll content in su- garcane seedlings using spectral images. Engenharia Agrícola, 41(4):475–484. https://doi.org/10.1590/1809-4430-Eng. Agric.v41n4p475-484/2021
Paz-Kagan, T., Shmilovitch, Z., Yermiyahu, U., Rapaport, T., Sperling, O. 2020. Assessing the nitrogen status of almond trees by visible-to-shortwave infrared reflectance spectroscopy of carbohydrates. Computers and Electronics in Agriculture, 178:105755. https://doi.org/10.1016/j.compag.2020.105755
Souza, A. C. F., Lima, J. R. F. 2023. Comportamento dos preços de manga Palmer ao produtor do Vale do Submédio São Francisco. Revista de Economia e Sociologia Rural, 61(1): 1-20. https://doi.org/10.1590/1806-9479.2021.250161
Souza, de A. P., Ferreira, I. J. S., Costa, D. dos S. 2022. Determination of quality atributes and ripening stage using Vis-NIR spectroscopy in intact seriguela and umbu fruits. Revista Engenharia na Agricultura, 30: 127-141. https://doi.org/10.13083/reveng.v3O-0i1.12929
Souza, I. C. O., Maia, G. A. M., Almeida. N. M., Abreu Neto, J. C., Freire, G. S. S. 2020. Sedimentary dynamic and composition of a tidal channel in a tropical hot semi-arid environment, NE Brasil. Anuário do Instituto de Geociência – UFRJ, 43: 144-155. https://doi.org/10.11137/2020_4_144_155
Notas

